Marqueurs SSR (microsatellites) et sélection assistée par marqueurs (MAS) chez la tomate affectée par le mildiou. Examen


Les marqueurs SSR (microsatellites), basés sur la PCR, peuvent être utilisés pour établir les cartes génétiques (ici cas de la tomate). Ceci permet la sélection assistée par marqueurs pour le trait de la résistance au mildiou.
Examen de rattrapage de l'élément du module 'Génétique et Amélioration des plantes' (partie 'marquage moléculaire et amélioration des plantes'), module 'Biochimie, Génétique et Amélioration des Plantes' du Parcours S5 : 'Biologie Appliquée aux Productions Végétales', Février 2013, Durée : 30 minutes .......... Corrections brèves à la fin de l'épreuve
QUESTIONS
Le mildiou de la tomate est une maladie cryptogamique causée par Phytophthora infestans, responsable de pertes énormes sur les productions des solanacées cultivées (pommes de terre, tomate , aubergines, poivron).

Plusieurs
essais de croisements dirigés réalisés entre la tomate cultivée (Lycopersicon esculentum) et des espèces sauvages, ont été réalisés dans le but d'introduire des gènes de résistance dans de nouvelles plantes qui seront testées par infestation expérimentale.
Afin de réaliser la cartographie génétique des traits de resistance au mildiou et des marqueurs moléculaires de types microsatellites (SSR), Zhu et et al., 2006 (Agr. Sci. China 5, 517-521) ont étudié la ségrégation du trait de la résistance au mildiou et huit marqueurs de types SSR, dont un correspondant à l'amorce TOM236 (marqueur TOM236). Pour atteindre ce but, les chercheurs ont suivi la ségrégation des traits de résistance et des marqueurs sur une population F2 provenant
d'un croisement de plantes de la population F1, elles mêmes dérivant
d'un croisement entre un génotype mâle résistant
au mildiou (CLN2037E, parent P1) et une variété de tomate
sensible (parent femelle, P2). Le test de la resistance des plantes
a été réalisé selon deux méthodes
différentes, dont l'une consiste à cultiver les plantes
dans un champs infecté par Phytophthora infestans (trait de la
resistance Ph-RoF) et l'autre correspondant à une incubation
des feuilles dans un milieu liquide contenant le champignon (trait de
la resistance Ph-RoL). Des tests préalables ont montré
que le caractère de la résistance au mildiou est dominant
sur le caractère de la sensibilité.
L'ADN a été purifié à partir de toutes les
plantes P1, P2, et F2 a subit une amplification par PCR (polymérisation
en chaîne de l'ADN) en présence d'amorces spécifiques
pour un marquage de type SSR. Les fragments d'ADN obtenus ont été
séparés par électrophorèse sur gel d'agarose
2,5% (p/v) et visualisés par le Bromure d'éthidium. L'électrophorégramme
relatif aux marquage SSR résultant de l'utilisation de l'amorce
TOM236, est représenté par la figure 1 (M indique les marqueurs de la taille de l'ADN, S et R renseignent sur la sensibilité et la résistance des plantes au mildiou, séparément).

L'étude de ségrégation du marqueur moléculaire TOM236 de type SSR et des traits de resistance au mildiou ROF et ROL a permis de montrer qu'ils appartiennent au même groupe de liaison, avec des distances génétiques (exprimées en cM) indiquées dans la figure 2.
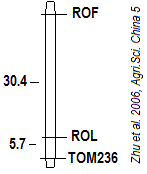
-- 1) Rappeler le principe de séparation de l'ADN par électrophorèse et la mise en évidence (visualisation) des fragments d'ADN par le bromure d'éthidium.
-- 2) Le marquage moléculaire de type SSR est basé sur la PCR. Rappeler les ingrédients requis pour la polymérisation de l'ADN (étape de l'élongation).
-- 3) Rappeler les origines du polymorphisme de type SSR (microsattelites).
Quelles en sont les caractéristiques principales
-- 4) A l'aide d'un tableau, comparer les marqueurs moléculaires
de tpe SSR et RAPD (nature des extraits d'ADN, étapes principales,
enzymes utilisées, effet de l'environnement, reproductibilité,
déterminisme de dominance-codominance).
-- 5) Pourquoi a-t-on souvent recours aux croisements par des
espèces sauvages dans des programmes d'amélioration génétique
de la tomate.
-- 6) En s'aidant de la figure 1, déduire la différence
structurale susceptible d'expliquer la différence de migration
entre l'ADN amplifié des plantes sensibles et résistantes
au mildiou. Comparer les génotypes à base des marqueurs
SSR, obtenus avec l'amorce TOM236 et montrés par la figure chez
les plantes de tomates, y compris les parents P1 et P2 (nature homozygote,
hétérozygote).
-- 7) A l'aide de la figure 2, préciser les utilisations
propices du marqueur moléculaire TOM236 de type SSR dans l'amélioration
génétique de la tomate pour la résistance au mildiou.
Justifier votre réponse.
CORRECTIONS BREVES
Revenir aux questions
Réponse à la Question 1: Principe
de séparation de l'ADN par électrophorèse et mise
en évidence (visualisation) des fragments d'ADN par le bromure
d'éthidium.
L'électrophorèse
de l'ADN est basée essentiellement sur le critère de la taille. La charge électrique négative de l'ADN permet
une migration vers l'anode (+). Parfois le forme de l'ADN intervient
dans la séparation, lorsqu'il existe plusieurs formes (circulaire,
relachée, superenroulée). La révélation
de l'ADN par bromure d'éthidium (BET) exploire la propriété
d'intercalation de ce dernier entre les deux brins d'ADN.
La. Lien utile : Electrophorèse
Réponse à la Question 2: PCR: ingrédients requis pour la polymérisation de l'ADN (étape de l'élongation).
Le mélange réactionnel de la polymérisation de
l'ADN contient l'ADN matrice (template), l'amorce (primer), les désoxyribonucléosides
triphosphates (dXTP = dATP, dCTP, dGTP et dTTP), la Taq DNA polymérase
et Mg++ (--> QCM
réplication ADN).
Réponse à la Question 3: Origines du polymorphisme de type SSR (microsattelites). Quelles en sont les caractéristiques principales.
Les microsatellites (SSR) sont constitués de séquences de di-, tri- ou tétra-nucléotides répétés en tandem. Le polymorphisme de type SSR repose sur la variation du nombre d'unités de répétition constituant le microsatellite. Parmi les caractéristiques principales, il y'a le multiallélisme. Lien: Marqueurs SSR (microsatellites).
Réponse à la Question 4: Comparaison des marqueurs moléculaires de tpe SSR et RAPD (nature des extraits d'ADN, étapes principales, enzymes utilisées, effet de l'environnement, reproductibilité, déterminisme de dominance-codominance).
| SSR | RAPD | |
| Nature des extraits de l'ADN | ADN hautement purifié | ADN partiellement purifié |
| Etapes principales | Amplification de l'ADN en présence d'une paire d'amorces (20-40 nucléotides) et de la Taq DNA polymérase, électrophorèse sur gel de polyacrylamide, révélation par une méthode appropriée. | Amplification de l'ADN à l'aide d'une amorce aléatoire de 10 nucléotides en présence de Taq DNA polymérase, électrophorèse sur gel d'agarose, révélation par une méthode appropriée comme celle utilisant le bromure d'éthidium. |
| Enzymes utilisées | Taq DNA polymérase | Taq DNA polymérase |
| Effet de l'environ nement | Non | Non |
| Reproducti bilité | Elevée | Moyenne |
| Caractère du marqueur (détermi nisme) | Codominant. Les individus hétérozygotes se distinguent facilement des parents | Dominant. Les individus hétérozygotes difficiles à distinguer des parents |
| Temps de réalisation | long | Rapide |
Lien
utile: Marqueurs
RAPD et SSR
Réponse à la Question 5:
Raisons du recours aux croisements par des espèces sauvages dans des programmes d'amélioration génétique de la tomate.
Réponse
à la Question 6: Différence
structurale susceptible d'expliquer la différence de migration
entre l'ADN amplifié des plantes sensibles et résistantes
au mildiou. Comparer les génotypes à base des marqueurs
SSR, obtenus avec l'amorce TOM236 et montrés par la figure chez
les plantes de tomates, y compris les parents P1 et P2 (nature homozygote,
hétérozygote).
La détail structural susceptible d'expliquer la différence
de migration entre l'ADN amplifié des plantes sensibles et résistantes
au mildiou réside dans le nombre de répétition du microsatellite. Les plantes résistantes sont connues par un nombre de répétition faible (fragments d'ADN migrant rapidement vers l'anode, car de taille faible).
Réponse
à la Question 7:Utilisations propices du marqueur moléculaire TOM236 de type SSR dans l'amélioration génétique de la tomate pour la résistance au mildiou.
Le marqueur moléculaire TOM236 de type SSR peut être utilisé dans la sélection assistée par marqueurs (MAS) appliquée à la résistance de la tomate au mildiou.
La cartographie génétique est basée sur l'observation
de la transmission des caractères héréditaires
(fréquence des recombinaisons des gènes). Les distances
génétiques sont le reflet de la fréquence de recombinaison.
En effet, le trait de la résistance au mildiou (comme testé
par incubation des feuilles en présence du champignon), noté
ROL, est lié au marqueur TOM236. Les deux marqueurs (morphologique
et moléculaire) ne sont distants que de 5,7 cM (très peu
de crossing over).
Lien: Cartographie génétique et MAS
LIENS UTILES
- BIOTECHNOLOGIES. TEST DES CONNAISSANCES
- Modes de reproduction des plantes (autogames, allogames)
-Reproduction asexuée
- Polyploïdisation, Haploïdisation des plantes
- Amélioration génétiques des plantes autogames
- Amélioration génétiques des plantes allogames
- Sélection Assistée par Marqueurs (MAS)
- Autogames, allogames. Exercice
- Sélection des plantes autogames. Exercice
- Sélection des plantes allogames. Exercice
- RFLP et sélection des protoplastes chez l'oranger. Examen S5_2012
- RAPD et sélection de la tomate. Examen S5_2012
- Microsatellites (SSR) et MAS chez la tomate. Examen 2013
- Microsatellites. Détection d'hybrides orangier (Citrus sinensis) x mandarinier (Citrus reticulata). Contrôle de rattrapage 2017
-Isoenzymes. Mutations
-Isoenzymes. Mutations
- Isozymes. Principe de détection
- Isozymes: loci, allèles
- RFLP
- RAPD
- AFLP
- SSR (microsatellites)
ENSEIGNEMENT DE MASTER
- Cours du Master Biotechnologies et Amélioration des Plantes,
Marrakech
- Travaux pratiques Master Biotechnologies et Amélioration des Plantes, Marrakech
- Master course 'Plant resistance against pathogens'